时间:2018年1月14日 天气:晴
Author:冬之晓
Email: 347916416@qq.com
今天是周日,我一早来到实验室,但是并不是搞科研,主要是过来进行博客的整理,最近我发现github里面可以使用jekyll这个框架
生成的静态HTML语言进行博客的撰写,非常开心,最近我就在把我以前的日志迁移到github的io上面。
这样一直弄到了晚上。这时,一起上英语课的杨瑞同学问我一个问题,她这两天刚好生病了,所以我决定帮帮她,最后我解决了这个
问题,感觉挺有意思的,下面详细介绍!
Python常用数据挖掘的工具包
python对于数据处理非常有好的语言,比如常用的scikit-learn和scipy 都可以用来进行机器学习和数据挖掘。同时为了使得结果可视化,Python还提供了非常好用的可视化工具包matplotlib和 seaborn。
使用Python进行层次聚类
聚类对于机器学习和数据挖掘来说都是一个非常常用的的工具。其中层聚类又一起显示效果和可解释效果好而在数据处理中非常常用,那是其缺点就是所需的 时间复杂度———和空间复杂度———比较高,但是对于少量数据来说无疑是最好的选择。
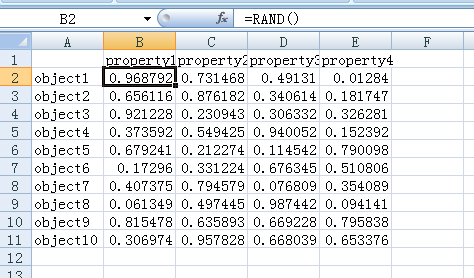
我们首先用excle的随机函数RAND()生成一个随机数据列表,进行测试,取名为test.xlsx,表格内容如下图所示:
然后就可以开始写代码了,首先需要导入要用到的包:
import pandas as pd
import seaborn as sns #用于话热图的工具包
from scipy.cluster import hierarchy #用于进行层次聚类,话层次聚类图的工具包
from scipy import cluster
import matplotlib.pyplot as plt
from sklearn import decomposition as skldec #用于主成分分析降维的包
1. 使用pandas里面的文件读取函数进行
pandas工具包是Python语言中专门进行数据结构化存储和数据分析的工具包,很多其他包都以pandas的结构化数据作为其函数的
输入,因此数据处理的第一步大多使用pandas进行数据结构化存储,关于excel文件,pandas就要专门的读取函数read_excel
,根据函数的说明文档,我们写下如下读取文件的代码
df = pd.read_excel("test.xlsx",index_col=0) #index_col=0指定数据中第一列是类别名称,PS:计算机程序一般从整数0开始计数,所以0就代表第一列
#df = df.T #python默认每行是一个样本,如果数据每列是一个样本的话,转置一下即可
这样里需要注意,后面使用各种Python的数据挖掘或者机器学习算法包时,默认每一行代表一个样本, 我生成的excel测试数据满足这个要求,因此不用改变,如果你读取的excel数据每一列是一个数据的话 需要先将数据转置一下,才能进行后面的操作
2. 绘制层次聚类图
读取数据后,我们就可以进行层次聚类图的绘制了,首先进入scipy参考文档页面,然后
找到聚类包Clustering package (scipy.cluster)->层次聚类scipy.cluster.hierarchy,在当前页面
下,就可以看到所有层次聚类相关的函数了,我们找到可以画图的函数dendrogram,进入该函数的文档页面
,发现该函数需要传入的第一个参数是linkage矩阵,这个矩阵需要函数linkage,进入该函数的文档页面
我们看到linkage的说明文档上面的函数scipy.cluster.hierarchy.linkage(y, method='single', metric='euclidean', optimal_ordering=False),
传入第一个参数是需要进行层次聚类的数据,这里即可用使用开始读取的数据变量df,第二个参数代表层次聚类选用的方法,底下罗列了七种方法,比如:
single方法代表将两个组合数据点中距离最近的两个数据点间的距离作为这两个组合数据点的距离。这种方法容易受到极端值的影响。 两个很相似的组合数据点可能由于其中的某个极端的数据点距离较近而组合在一起。 即两个簇之间的距离使用公式: 计算。complete方法代表与Single Linkage相反,将两个组合数据点中距离最远的两个数据点间的距离作为这两个组合数据点的距离。 Complete Linkage的问题也与Single Linkage相反,两个不相似的组合数据点可能由于其中的极端值距离较远而无法组合在一起。 即两个簇之间的距离使用公式: 计算。average方法代表是计算两个组合数据点中的每个数据点与其他所有数据点的距离。将所有距离的均值作为两个组合数据点间的距离。这种方法计算量比较大,但结果比前两种方法更合理。 即两个簇之间的距离使用公式: 计算。weighted即两个簇之间的距离使用公式: 计算。centroid即两个簇之间的距离使用公式: 计算。median同centroidward即两个簇之间的距离使用公式: 计算。
第三个参数代表距离计算的方法,即上面方法中的dist()函数具体的计算方式,具体方式可以见这个页面。
然后这里我随便选择两个,然后将返回的结果Z传入dendrogram函数,代码如下:
Z = hierarchy.linkage(df, method ='ward',metric='euclidean')
hierarchy.dendrogram(Z,labels = df.index)
运行程序,即可得到层次聚类图如下:
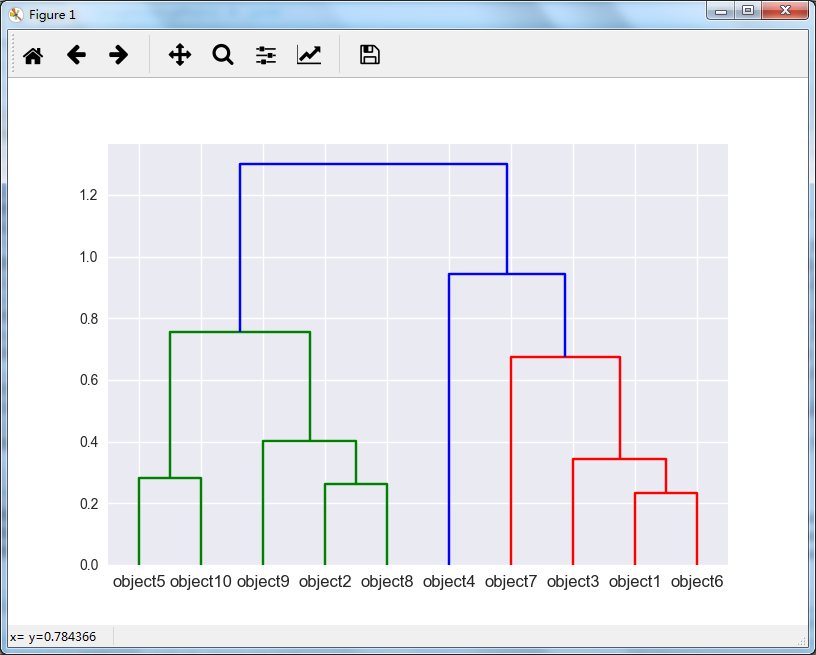
根据上图,我们即可看到在不同的位置裁剪即可得到不同的聚类数目。但是我们具体要聚集多少类呢? 我们先写上裁剪的代码:
label = cluster.hierarchy.cut_tree(Z,height=0.8)
label = label.reshape(label.size,)
上面代码中的高度我取的是0.8,根据层次聚类图明显是聚成三类,为啥我要这样取值呢?下一小节给出答案。
3. 绘制两个主成分方向坐标的散点图
为了将聚类结果可视化,我们需要降维,因为在大多数情况下,我们处理数据的维度超过三维,因此可以使用主成分分析法,
找到占据方差最大的两个维度的散点得到,然后进行绘图,来观察结果。
由于我没有在scipy里面找到主成分分析的相关函数,因此就在scikit-learn包里面找相关函数,发现里面有主成分分析的函数PCA,进入该函数的说明文档
,我们可以进行相关操作和绘图,代码如下:
#根据两个最大的主成分进行绘图
pca = skldec.PCA(n_components = 0.95) #选择方差95%的占比
pca.fit(df) #主城分析时每一行是一个输入数据
result = pca.transform(df) #计算结果
plt.figure() #新建一张图进行绘制
plt.scatter(result[:, 0], result[:, 1], c=label, edgecolor='k') #绘制两个主成分组成坐标的散点图
for i in range(result[:,0].size):
plt.text(result[i,0],result[i,1],df.index[i]) #在每个点边上绘制数据名称
x_label = 'PC1(%s%%)' % round((pca.explained_variance_ratio_[0]*100.0),2) #x轴标签字符串
y_label = 'PC1(%s%%)' % round((pca.explained_variance_ratio_[1]*100.0),2) #y轴标签字符串
plt.xlabel(x_label) #绘制x轴标签
plt.ylabel(y_label) #绘制y轴标签
运行代码,得到下图:
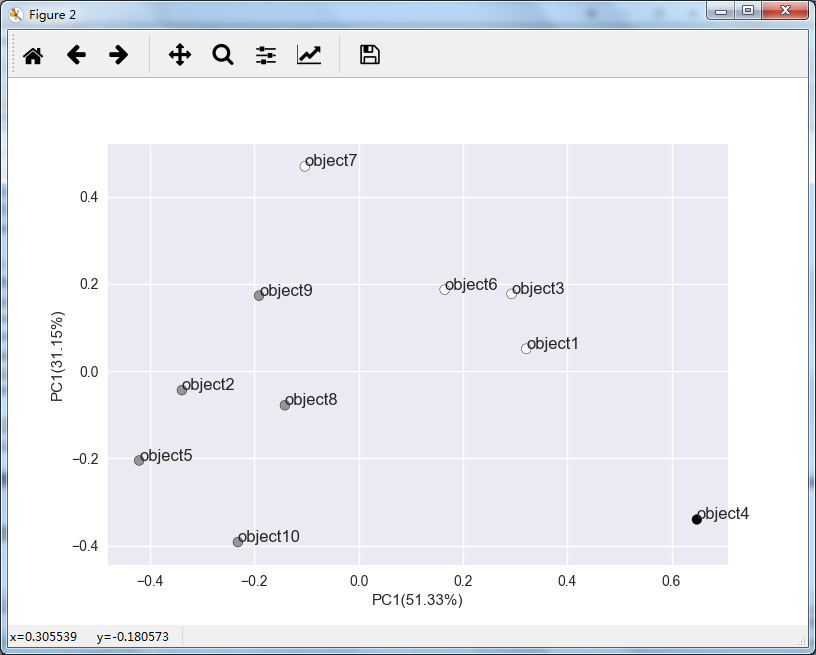
根据改图,我们可以看到大致分为三类比较合理,因此上一小节层次聚类裁剪的高度取了一个可以裁剪得到三类的高度0.8。
4. 绘制热图
热图的绘制非常简单,因为seaborn的工具包非常强大,我们使用clustermap函数即可,该函数的
说明文档
中有详细介绍,仅需一行代码,即可搞定,代码如下:
sns.clustermap(df,method ='ward',metric='euclidean')
运行代码,得到下图:
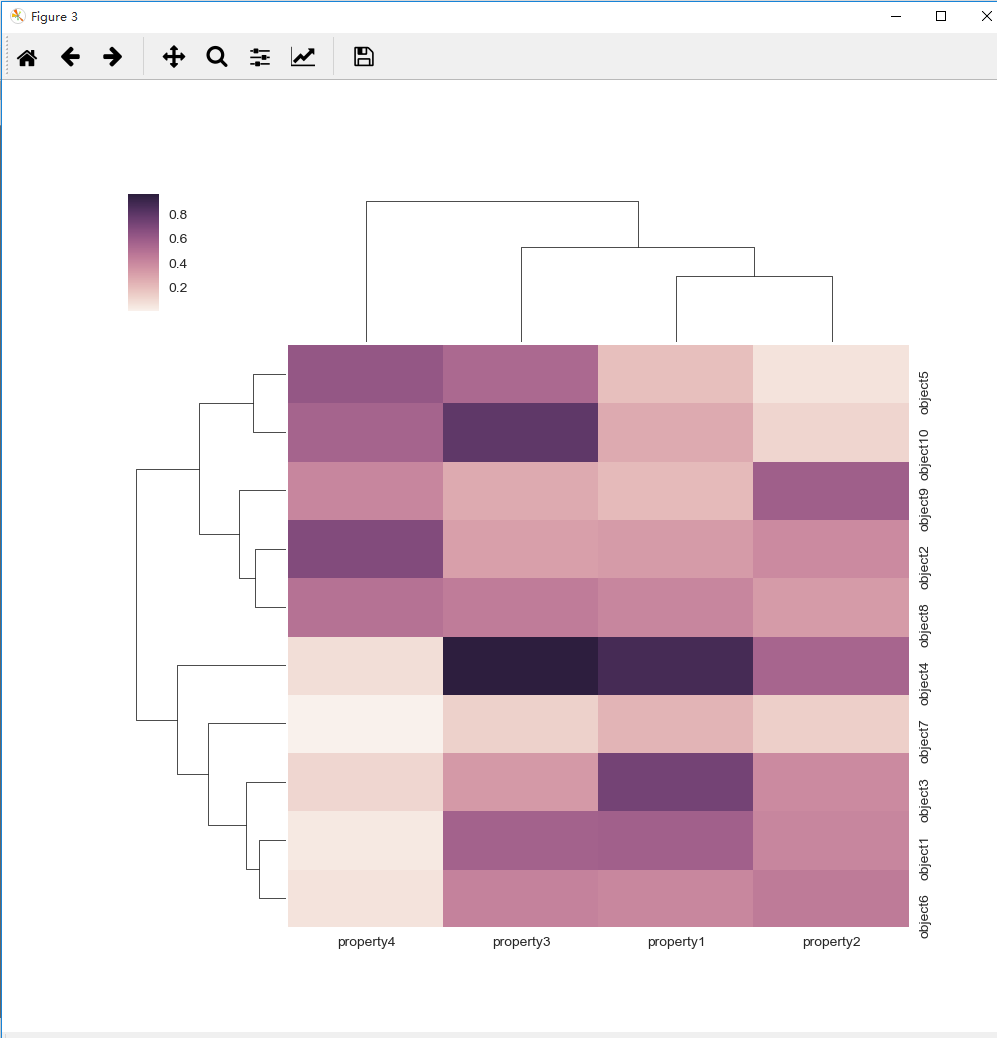
最后,本篇的全部代码在下面这个网页可以下载:
https://github.com/Dongzhixiao/Python_Exercise/tree/master/hierarchy